公众科学研究、大数据和人工智能——蛋白质组学研究 Foldit 的启示
知乎@咸球
原刊于 化术-知乎专栏
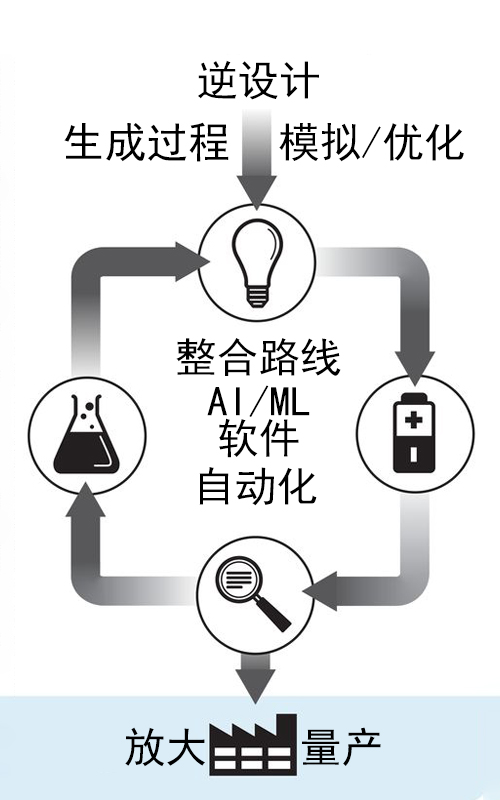
题图来自:**Science, 2018, 361(6400):360-365**(已翻译,有所修改)
摘要:以Chemputer为新兴代表的组合化学在自动化合成、测试方法中已初露锋芒,同时,近年来人工智能和大数据技术在化学研究中的应用亦悄然兴起,而以Foldit为代表的公众科学项目研究也在稳步发展。合成化学作为化学研究的基础,又将在新科技革命的大背景下具有新的变化,“材料4.0”也正成为合成化学研究的远景趋势。未来,大数据支撑的社会生产将前所未有地将生产、研发乃至使用者紧密地联系在一起。标准化的数据库和蓬勃发展的数据挖掘产业终将为合成化学带来新的范式,低门槛、高参与度的公众科学研究也才将真正地变革科学技术。
引言
传统的合成化学多采用图 1所示的研究范式,也即通过设计材料、合成分子、制作装置原型、表征测试的循环进行研究,测试结果符合使用需求后进行放大量产。 [^1]这一范式中每一个步骤都可能需要数年时间完成,致使材料研发工作的整体进度严重落后于材料需求的提出。而耗时如此之长的研究路线也在一定程度上导致了研究小组或研究机构的信息闭塞和不互通。目前已有一些研究人员意识到了这一系统性问题,不过相关的研究范式的调整仍有相当长的路要走。
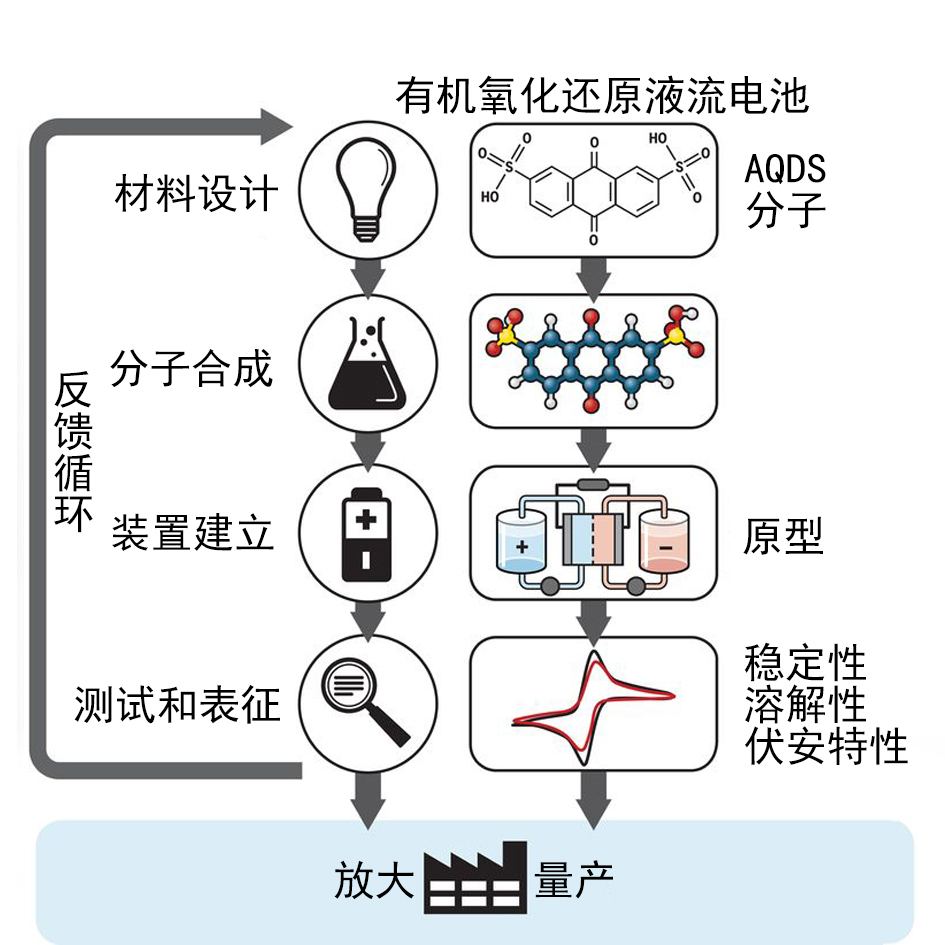
图 1 传统材料研究的范式 [^1](以有机氧化还原液流电池材料 AQDS 为例)
公众科学研究是指研究者将经过简单加工的科学问题发布给公众进行协助研究,具有门槛低、科普性强、直观可视化等特点。其对于生态学、环境科学等领域的大规模研究起到了相当重要的作用[^2],也在分子生物学等领域,形成了以 Foldit 和 Rosetta 系列项目为代表的公众参与的创新性研究模式。借助公众科学研究的方法,对合成化学乃至整个化学研究有可能带来新的研究范式。
公众科学研究的典型代表 Foldit(图 2)是一款由华盛顿大学(University of Washington)等机构在 2008 年起开发维护的基于 Rosetta 平台的在线蛋白质结构预测游戏,目前其用户 ID 数已达 75 万以上。[^3][^4]其计算模型是基于粗粒化模型的 Rosetta 结构预测方法,主要借助游戏者的操作进行构象搜索获得最低的“Rosetta 分数”。[^5]借助 Foldit,研究者可以高效地获得大量数据,甚至是在传统的计算搜索方法中需要极长时间才能得到的计算结果,如解决 M-PMV 逆转录病毒蛋白酶晶体结构,更重要的是研究者可以通过研究游戏者解决问题的方法和步骤改进现有软件的算法。[^6]Foldit 在蛋白质组学的应用一定程度上得益于高通量蛋白质组学实验的数据积累和数据模型。而在材料和合成化学领域,高通量的方法也有一些新的研究趋势。

图 2 Foldit 游戏首页的说明图片[^3]
(图中写道:“点此了解您如何通过玩 Foldit 为科学做贡献”)
对比环境科学和蛋白质组学的公众科学研究项目,若要将公众科学研究运用到材料合成等化学领域基础研究中,主要需要解决优质轻量的粗粒化模型的建立和合适的人机交互模式与数据收集两个问题。目前在化学信息学基础上,基于机器学习技术发展出了一系列新的模型和方法,人工智能技术和相关产业对于粗粒化模型建立问题的解决具有相当的潜力。
大规模的高性能计算系统使人工智能用于基础科学研究和材料等大数据分析成为可能。其中,以连接主义为主的神经网络技术最引人注目,这得益于反向传播算法的成熟和大量科技企业的资金投入。各类语言、游戏和机器视觉研究项目使得机器学习在近年来获得了长足发展和公众知名度。人工智能技术在化学领域的应用最早可追溯至有机合成化学逆合成分析的研究,逆合成分析及其计算机程序其实就是早期符号主义人工智能指导下诞生的的专家系统。而Sanchez-Lengeling等也提出了基于人工智能、软件系统的闭环材料逆向设计范式(图 3)。[^1](其将材料工程的设计、合成、原型和测试等步骤发展为人工智能系统协调辅助的体系化动态系统)本文还将对合成化学和人工智能结合形成交叉学科的研究现状进行简要介绍。

图 3 “闭环”的材料工程新范式 [^1]
高通量化学合成与测量
Foldit的理论基础来源于大量蛋白质组学的实验研究数据的积累,而即便引入了包括模拟和化学信息学等数据分析手段,实验仍是化学研究的基本手段。不过随着材料研究的深入和对于定制化材料的需求不断上升,传统长周期的材料合成以及人工穷举式的合成方法难以满足研究的需要。以自动化合成和新型高通量合成为代表的组合化学研究方法应是未来材料设计和研发的前沿。基于自动化合成和高通量技术的材料研究所具有的快速、批处理和标准化的优势是传统的材料设计周期望尘莫及的,同时其又能积累大量高水平、高可靠性和标准化的材料数据参与图 3所示的“闭环”研发过程。自动化合成更能将研究人员和实验人员的精力从重复性劳动中解放出来,促进更多创造性成果的产生。
组合化学:多肽的固相合成
Bruce Merrifield因为“他在固体基质上化学合成方法的发展”获得了1984年的诺贝尔化学奖。[^7]Merrifield的关键贡献在于开发了通过将肽链固定在固相上以减少氨基酸聚合产物中大量杂质的方法。类似的方法也已被用于其他蛋白质、低聚物、小分子和寡糖的制备中。[^8]这类组合合成的作用在于其能一次性平行地合成得到大量可供测试的目标分子,这极大地减少了时间成本和原料消耗。
Chemputer:自动化合成
Chemputer(图 4)是Steiner等设计出来进行复杂有机分子自动化合成的系统。其包括一个模块化的机器人系统和用于控制机器人模块的“化学程序设计语言”。这一系统将分子的组装合成抽象为用程序设计语言控制的合成步骤,且不需要过程中的人工干预,就可合成得到可他敏(盐酸苯海拉明)、卢非酰胺和西地那非。[^9]这一系统的核心是将化学合成的过程抽象为可用程序设计语言表达的无二义性的过程。除药物分子有机化合物外,其也可能适用于在流体条件下制备的纳米材料或相应前驱体。
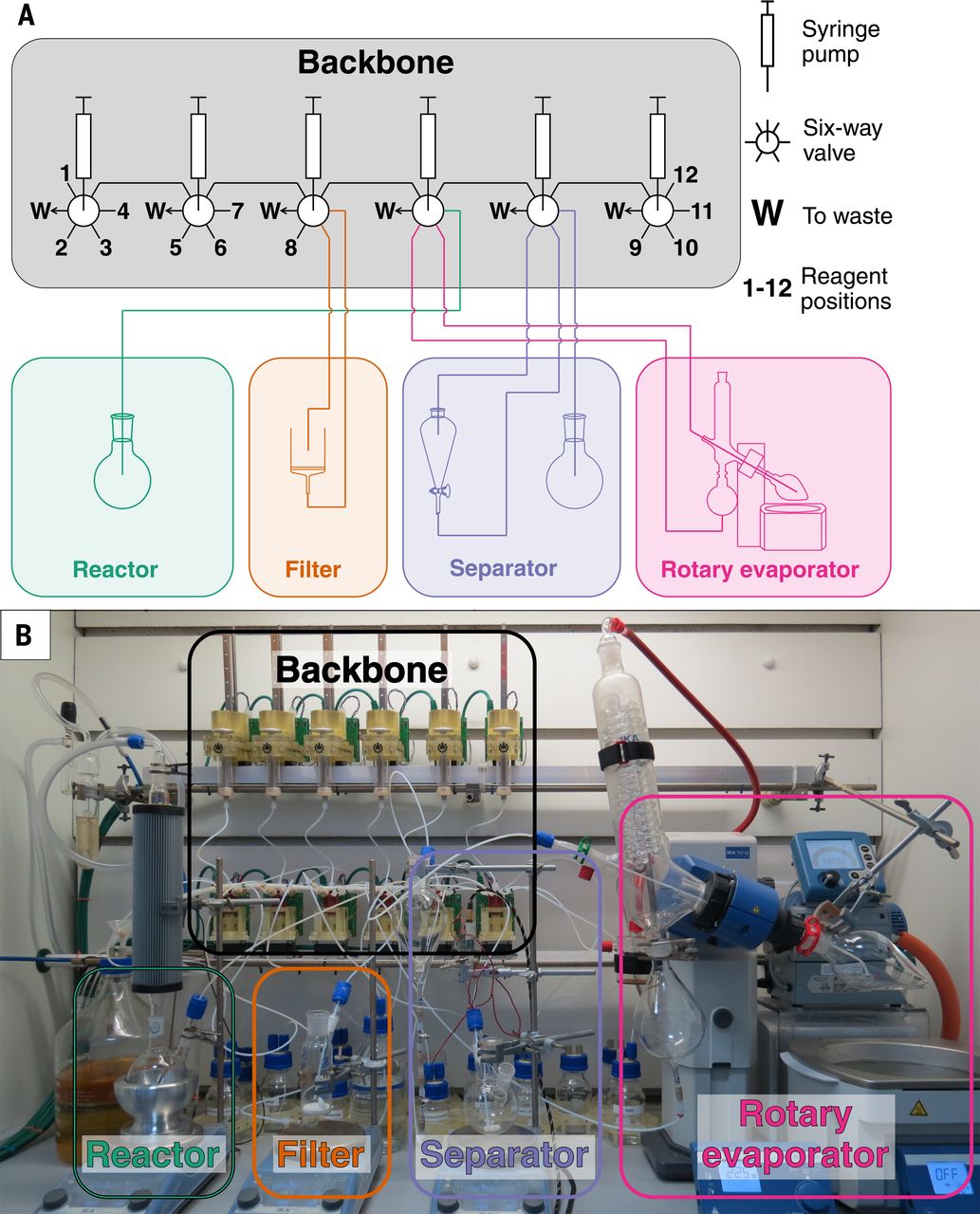
图 4 Steiner 等设计的 Chemputer 结构图(A)和实物(B) [^9]
Chemputer系统的亮点在于其采用的化学程序设计语言可直接迁移到采用相同机器人装置的其他Chemputer上,因此带来的标准化和自动化的流程对于产生高可靠性数据和标准化制备数据具有重要的意义。
材料高通量合成与测试
高通量方法目前仍是功能分子和材料研究中一项重要的实验技术,其可用于功能分子的合成、仿生合成、聚合物材料的组合合成等平行合成,本节结合研究实例主要介绍高分子和合金材料的高通量合成。
高分子化合物的高通量合成与测试
高分子化合物一般需要在溶液体系中合成,使用高通量方法比较难度较大,不过基于合适的负载体和良好的分离、混合过程,亦能实现一定数量的高分子化合物的高通量合成。而组合化学的高通量合成侧重于在聚合物中产生梯度、表面分布乃至使用喷墨印刷等技术。[^10]合理的参数和合成技术控制甚至可以实现高分子浊点的可视化(例如图 5),抑或是嵌段共聚物嵌段长度对其溶剂效应的影响[^11][^12]。可以发现高分子的高通量合成大多依托于流体化学的技术,主要是通过流速保持层流、保持浓度梯度进而控制高分子产物的组成。

图 5 Meredith 等制备得到的温度/组成正交梯度的聚苯乙烯/聚乙烯基甲基醚膜的最低共溶温度(LCST)浊点“曲线” [^12]
金属合金材料的高通量合成
金属合金材料的高通量实验相对成熟,其中美国国家标准与技术研究所(NIST)和国家可再生能源实验室(NREL)还针对合金材料的高通量实验(high-throughput Experimental, HTE)提出了标准化的高通量测试协议。相关的方法也有助于材料基因组计划(The Materials Genome Initiative)的研究,如Green等对基于HTE的多种金属材料研究的综述回顾,分析指出HTE方法的可迁移性、标准化等问题,也展望了将HTE体系与教育、大学产业开发应用等结合的思路(表 1)。[^13]他们所指出的允许公众访问参与HTE材料虚拟实验室(HTE-MVL)的想法也正是公众科学研究这一范式的具体形式。
表 1 Green等对HTE用于加速材料发现和商业化的建议[^13]

针对相关问题,Hattrick-Simpers等还以Zn-Sn-Ti-O赝三元氧化物材料为例,对不同实验室中的高通量组合实验技术的实验结果做了比较,验证了高通量实验材料合作组织(HTE-MC)间进行样品和数据交换的概念可行性。[^14]相关研究者在不同实验室之间进行的方法对比和数据交换体系验证其实与公众科学研究中所采用的数据交换协议及相关模式有很高的相似性。
小结
除上述固相合成、金属合金高通量合成外,材料高通量合成还可与新兴的机器学习等手段结合,如Pardakhti等结合机器学习与组合合成技术对金属有机框架(MOFs)材料进行结构和化学性质分析,并预测相应材料对于甲烷的吸附性能。[^15]材料高通量合成最重要的特点在于其可以一次性生成大量的数据供分析使用,特别是具有标准化合成测试手段和标准化数据存储系统的高通量合成技术,这是合成化学研究的新范式的重要基础。
化学信息学和计算化学
化学信息学
化学信息学(Cheminformatics,曾称作Chemoinformatics),是对于化学信息资源的整合、数据转化和应用于药物导向识别、优化设计等的一门科学。[^16][^17]自上世纪计算机运算能力显著提高和数据库信息系统的大规模应用起,对于化学信息资源的研究才逐渐发展成为一门新的学科。按照化学信息资源的处理过程分,化学信息学主要包括化学数据的收集与存储,以及化学数据的分析两个大的研究领域。[^18]其对于化学研究数据的标准化存储、展现和分析具有相当重要的意义。
化学数据分析与存储,可以细分为化学信息的检测、数字化、数据库设计和化学信息的计算机表示等。[^18]其中化学信息的检测即和仪器分析化学中的各类检测手段密切相关,对相应仪器记录信息的数字化存储主要研究相应信号器的记录和固定化,更多的是信息科学和信息工程的研究对象。而具体的数据库信息表示是化学信息学对数据收集和存储研究的主要内容。化学信息的计算机的成熟表示有XML(扩展标记语言,Extensible Markup Language)、SMILES(简单分子线性输入规范,Simplified molecular input line entry specification)、由国际纯粹与应用化学联合会和美国国家标准技术研究所(National Institute of Standards and Technology,NIST)联合制定的国际化合物标识InChI(International Chemical Identifier),此外还有化学文摘服务社的CAS登录号以及分子图形学表示的研究等。
而在化学数据分析领域,有定量构效关系、计算机辅助结构解析、分子模拟和计算化学、人工智能算法等研究工具。[^18]药物化学中对于分子生物活性的预测,最早是通过Hammett方程[^19]、Hansch方程[^20]等建立活性与分子的电性、立体参数和疏水性的回归分析模型,在不断修正完善中形成了定量构效关系(Quantitative Structure - Activity Relationship,QSAR)。而计算机辅助结构分析最著名的例子就是计算机辅助的逆合成分析(Computer-Assisted Retrosynthetic Analysis)[^21][^22],另外还有使用计算机对于分子的质谱裂分、核磁共振谱图、晶体衍射谱图进行预测和拟合的工具等,均是得益于计算机辅助结构分析的相关研究。
化学信息学的数据形式
标准化和规范化的数据形式和数据存储是合成化学数据集中必须的基础技术,但目前化学信息学的数据表示仍要根据不同的使用途径有所区别,且目前并没有非常统一的标准。而不同领域的数据表示也各有偏好。
化学物的表示
有机化合物的线型编码
线型编码主要用于表示简单线性结构分子或线性环系,比较著名的线型编码有WLN编码(Wiswesser Linear Notation)(如图 6),其通过字母代表官能团,运用数字和字母的组合表示有机化合物的化学结构;此类表示可以将多数复杂结构分子的信息完整地压缩存储到计算机系统中,且根据检索、人工智能识别等各类用途的需求,对无二义性和唯一性等特性各有侧重。检索和数据库登记中更多使用如和CAS号对应的唯一标识,相同分子有且只有唯一的编码。而在表达化学结构信息的人工智能识别系统中,更侧重于完整有效无二义性地反映分子的结构组成信息。
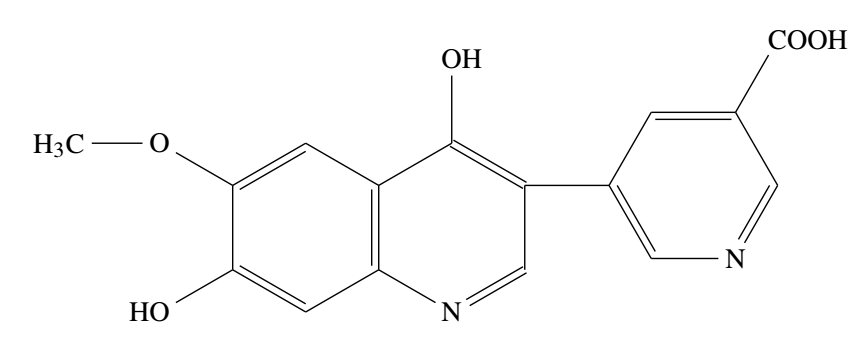
图 6 WLN 编码为 T66 BNJ EQ HO1 IQ D CT6NJEVQ 的分子[^18]
分子结构的可视化:分子图形学
分子图形学保留了分子结构的更多直观信息,常见的方式有分子建模、基于球棍模型的空间实填充模型(Solid space-filling,又称CPK模型)、蛋白质等大分子的带状模型、分子表面的概念等[^18],同时还有虚拟现实(VR)等新技术的应用。本文第1章提到的Foldit游戏和蛋白质建模工具[^23]就是使用蛋白质大分子带状模型等进行科学可视化的一个杰出成果。
理化性质信息和定量构效关系
基于分子描述符方法的分子信息数据除包含分子结构的信息外,还通过使用疏水性参数、电性参数、立体参数、拓扑类参数等描述分子结构特征信息。结合定量构效关系(QSAR)等方法,建立生物活性、毒性、药物代谢动力学等生物活性性质以及熔点、沸点等与结构特征信息的关系,可通过线性回归、支持向量机等模型建立相应的联系。
化学信息数据库和数据集
传统上的化学信息数据库主要存储分子的性质谱图等信息,以利合成化学中作为标准数据等进行参考;或是如SciFinder® [^24]等存储有机分子和相关的反应、合成方法信息等。
而目前包括SciFinder® 在内的合成化学产生的数据集仍是新兴领域,目前整理归纳文献报道实验的数据集有英国皇家化学会的chemspider[^25]、与Elsevier有合作的Citrine Informatics[^26]、施普林格·自然集团开发的Nature Nano数据库[^27]等。
计算化学与材料模拟
计算化学是广义的化学信息学的一部分,主要包括基于量子力学的量子化学模拟、基于分子力场的分子力学模拟和分子动力学(MD)模拟以及相应的谱图、性质模拟等。计算化学涉及的内容在材料建模模拟中[^28]主要是极小尺度和极短时间的模拟。(图 7)
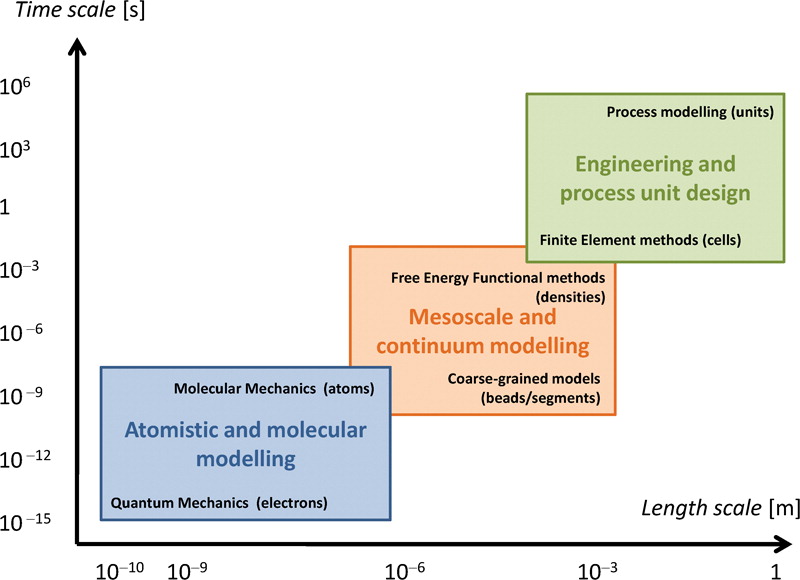
图 7 材料科学中各个尺度的建模和方法[^28]
从左至右依次为:原子和分子模型(包括量子力学(电子)和分子力学(原子))、介观和连续介质模型(包括粗粒化模型(液滴和分割)和自由能泛函方法(密集的))、工程和工艺单元设计(包括有限元和过程建模)
在图 1 的基础上,将材料科学的建模和计算方法纳入研究体系闭环中,则能够充分发挥计算机系统和大量数据在研究中的新作用。如材料计算工程(Integrated Computational Materials Engineering,ICME)来源于材料基因组计划,是将计算手段所获得的材料信息与产品性能分析和制造工艺模拟相结合,以期加速材料产品开发过程的工程体系。(如图 8 [^29])这一体系中充分地结合了化学的基础原理和计算机模拟技术,使研究人员在开发特定功能材料时能从更全面和更原理性的角度进行设计和开发。
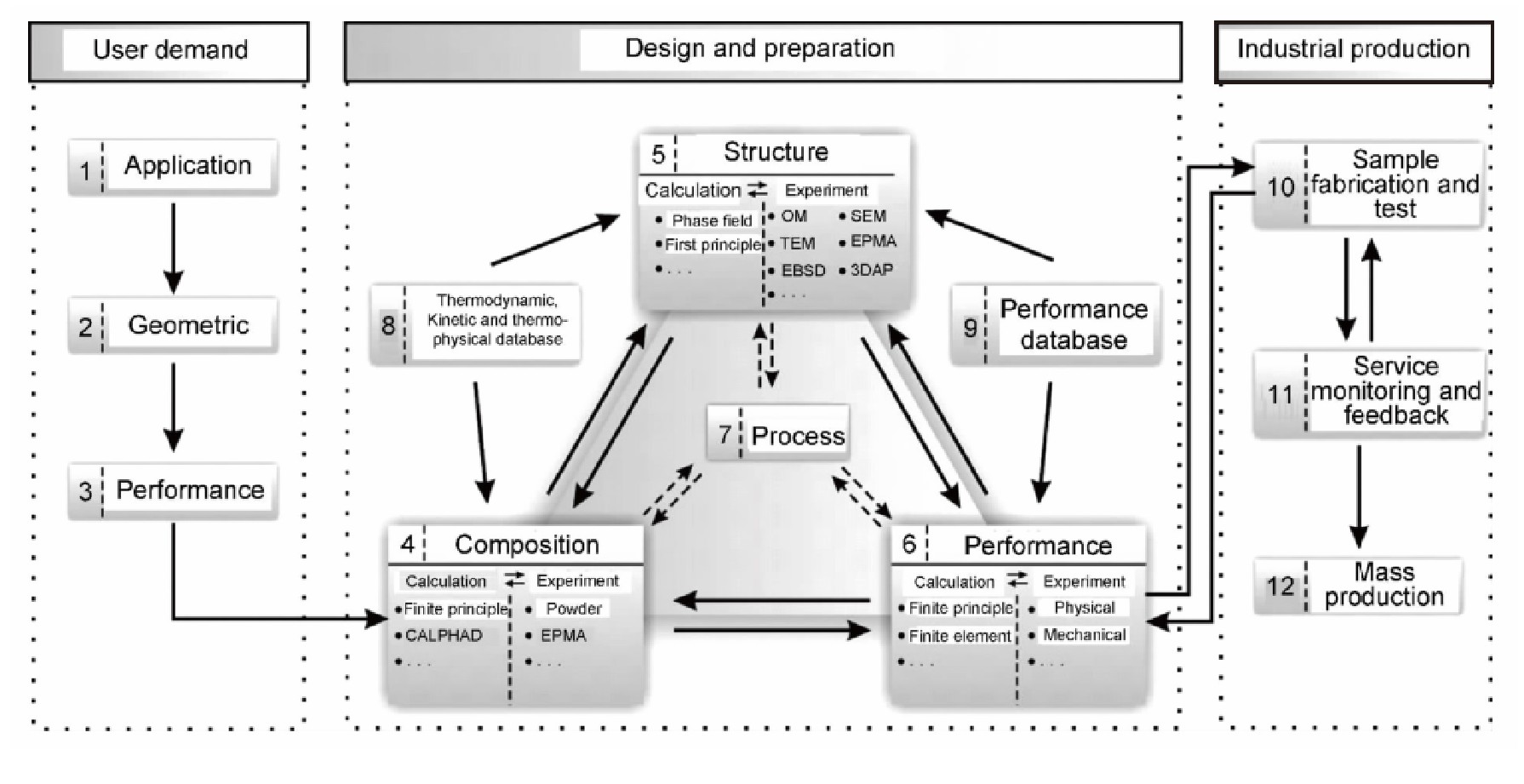
图 8 硬质合金研发过程中的集成计算材料工程(Integrated Computational Materials Engineering, ICME)框架 [^29]
标准化的分子和材料仿真技术是在大规模数据背景下相当重要的工程技术手段,近期亦有对于分子动力学模拟数据和相应计算条件数据集合的可复用性的探讨。[^30]在计算、模拟和合成化学大数据的背景下,更多的计算方法和数据集,以及在计算能力更强的计算机上使用更先进的算法和模型是在材料合成领域十分具有前景的。而且各类不同的化学信息学、计算化学、材料科学的模拟方法均在不同的尺度和不同的时间层次上有不同的发展需要和不可替代性,如果要取得进一步的研究成果和技术方法,在工业领域建立大型标准化和基准化的数据库和计算服务以供方法比较同样十分必要。[^31]
新范式:人工智能辅助和基于公众科学研究的合成化学
人工智能技术与化学
人工智能指智能机器所执行的通常与人类智能有关的智能行为,涉及学习、感知、思考、理解、识别、判断、推理、证明、通信、设计、规划、行动和问题求解等活动,有符号主义(基于物理符号系统假设和有限合理性原理)、连接主义(基于神经网络和仿生算法的连接机制等)、行为主义(基于控制论和感知-动作型控制系统)等不同方法和流派。[^32]
人工智能技术在化学的应用最早可追溯至有机化合物逆合成分析,其是符号主义人工智能在化学中的典型代表。[^21][^22]而近些年来随着以神经网络为代表的连接主义人工智能的迅猛发展,包括自然语言分析(NLP)、深度神经网络(DNN)及深度学习技术[^33]在化学中已颇具应用前景。
按照所应用的研究领域,化学研究中人工智能技术主要可分为应用研究中的实验数据分析模型和基础理论研究中的智能分类与计算模型。前者有专门的化工和热力工程化学研究(包括用于人工智能分析的标准化数据结构的推广[^31])。在后者,即基础理论研究中,包括专家系统等的人工智能技术在化学研究中应用比较早,如有机化合物结构解析的CHEMICS系统等。而神经网络、数据挖掘等近年来又呈现出新的应用前景,如基于药物分子指纹等结构信息预测分子性质等[^34]。化学信息学和组合化学主题的学术期刊目前也主要着眼报道一些人工智能辅助研究的报道,如ACS出版的《Journal of Chemical Information and Modeling》和《ACS Combinatorial Science》等,均有人工智能与合成化学应用的研究报道不断涌现。
机器学习和材料发现
“材料发现”其实是材料化学研究的根本目的,而材料4.0则是在工业4.0的大背景下,基于大数据革命和智能化工业生产的理念,将数据科学应用于材料开发生产的整个生命周期中,以促进合成工业和材料研发的快速迭代,并改善及解决现有的工业材料生产中的浪费、环保等问题的材料研究新理念。[^35]材料4.0相较于以前的“材料3.0”最大的革命性因素在于其对于数据的集约化、进一步挖掘和革命性运用。
在大数据革命和智能化工业的大背景下,材料研究领域本身需要做出诸多调整。就如传统工业在数字化和智能化浪潮中纷纷引入精准控制和细化对于产品全周期的把握,材料研发本身也应当考虑运用数据科学和智能化的技术手段。以人工智能为代表的数据科学手段已不是什么新鲜事,但其在合成化学中的应用仍有一定发展空间,如前文提到的标准化的数据交换格式、易于使用的应用程序接口[^13]和深度学习技术[^36][^37]乃至直观和可视化的操作界面等。[^35]
大数据材料发现研究的动态和趋势
Materials Project与PyMatgen
Materials Project[^38][^39]是加州大学伯克利分校材料科学与工程系副教授克里斯汀·佩尔森主导的开放、开源的材料数据库,截止2019年12月5日,其已有至少12万种无机化合物和53万种纳米孔材料等的实验或理论计算数据。[^38](如图 9)
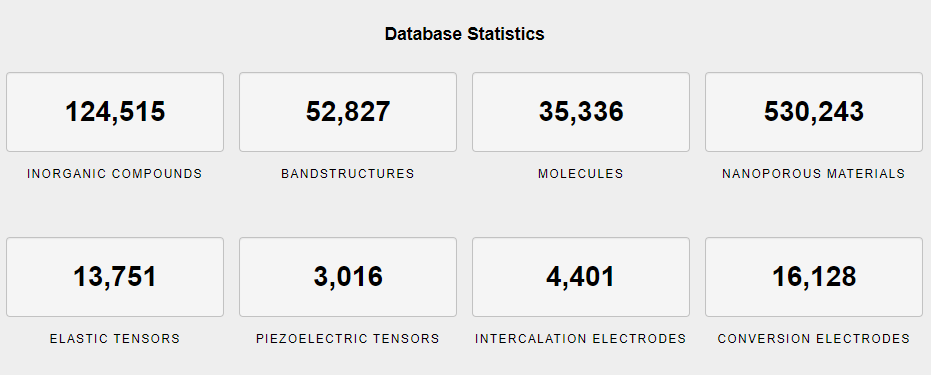
图 9 截止 2019 年 12 月 5 日 Materials Project 的数据规模[^38]
Materials Project基于PyMatgen[^40]等材料大数据分析平台,对于以json格式存储的数据文件,可以快速操作完成材料理化性质参数查询、理论计算能带绘制、相图、电化学(化学势)稳定性等的计算。而基于知识共享-署名 4.0 国际(CC BY 4.0)协议的Materials Project,极大的自由度更是赋予了大规模数据集的无限可能,因而也得到了美国能源局、美国国家科学基金会以及Gillette(吉利)、Volkswagen(大众)等的赞助。[^38]
Materials Project数据库中的材料信息可以直接通过其提供的API访问,且每个数据项目也有DOI直链,通过API和网页应用提供的能带、能级、稳定性乃至界面反应活性信息,研究者可以快速地筛选出与需求相关的材料的范围。
国家自然科学基金
本文撰写时2020年度国家自然科学基金项目指南尚未公布,但2019年项目指南[^41]即已对有机高分子材料学科提出“发展以高效‘理论指导-实验验证’为目标的高分子材料基因工程研究方法”、对于化学理论与机制也提出“鼓励发展基于系统原理的分子结构信息的存储、检索、变换和挖掘算法”、对于材料化学研究提出“关注利用人工智能优化先进材料的结构设计与制备过程”、对于化学工程与工业化学提出“鼓励化工大数据与智能过程”等涉及数据科学和系统科学的研究指导。
研究机构或企业
Citrine Informatics公司运作着一个数据量庞大的材料库平台,同时开发使用人工智能应用进行材料发现和优化制造的相关研究。[^26]而据称,中国科学院计算机网络信息中心材料基因工程信息技术应用实验室的MatCloud平台也有协助VASP使用者进行材料研究计算的想关应用。拥有大量文献资源的出版社,如前文提到的Nature Nano数据库[^27]同样在进行人工智能和材料发现相关的开发运营。
公众科学研究
科学研究工作归根结底要服务于社会,而不论是Foldit,材料4.0,人工智能技术对社会生活带来的巨大变化,还有是各类研究机构和企业对于材料数据等众多自然或人造数据产物的积极关注,都预示着即将到来的科技革命绝不仅仅是多了些新技术或者产品价格便宜一些那么简单。数据,而且是大规模数据和相应的应用技术才是未来科学研究的发展方向。而人工智能技术和更多的数据科学技术将像Foldit降低公众了解蛋白质组学研究的难度一样,也有希望降低公众对于数据量更庞大的材料科学和合成化学研究的理解难度。也如Quantum Moves这款游戏[^42]一般降低公众对于量子物理的理解难度类似,在大量的合成化学研究数据和应用技术的背景下,无论是应公众科学普及的需要还是专业人员对材料4.0数据规模化和可视化应用的需要,类似的具有公众科学研究性质的材料公众科学项目或许已然萌芽。
对于材料和合成化学公众科学项目,可以肯定的是其一定要基于大量可靠合成或理论研究数据库,通过基于人工智能等技术的粗粒化模型等可在个人PC上运行的小型软件,可能会运用虚拟现实(VR)或增强现实(AR)技术,让具有简单科学知识的受众通过简单的互动操作,“设计”或“组合”出新的分子、材料乃至装置。如Foldit的社交化运营一般,相应的也将产出大量的新研究思路,甚至新材料、新设计。同时还可能发挥如Chemputer一类自动化高通量生产工具的作用,让玩家在线合成自己想要的分子或材料,提供“个性化”的合成化学。当然,公众科学研究项目的成熟,必定意味着专业的研究人员拥有了更强大的研究工具,也正如Foldit与Foldit standalone的关系一样,将实现良性发展。
结语
标准化的数据库和蓬勃发展的数据挖掘产业终将为合成化学带来新的范式,低门槛、高参与度的公众科学研究也才将真正地变革科学技术。有朝一日,制约合成化学研究的将不再是研究经费、技术条件和时间,而仅是想象力。
参考文献
[^1]: SANCHEZ-LENGELING B, ASPURU-GUZIK A. Inverse molecular design using machine learning: Generative models for matter engineering[J/OL]. Science, 2018, 361(6400): 360–365. http://www.sciencemag.org/lookup/doi/10.1126/science.aat2663.
[^2]: SILVERTOWN J. A new dawn for citizen science[J/OL]. Trends in Ecology & Evolution, 2009, 24(9): 467–471. https://linkinghub.elsevier.com/retrieve/pii/S016953470900175X. DOI: 10.1016/j.tree.2009.03.017.
[^3]: Foldit Developers. The Science Behind Foldit | Foldit[EB/OL]. 2019[2019-11-25]. https://fold.it/.
[^4]: 中国分布式计算总站. Foldit - 中国分布式计算总站[EB/OL]. 2019[2019-11-25]. https://equn.com/wiki/Foldit.
[^5]: COOPER S, KHATIB F, TREUILLE A, et al. Predicting protein structures with a multiplayer online game[J/OL]. Nature, 2010, 466(7307): 756–760. http://www.nature.com/articles/nature09304. DOI: 10.1038/nature09304.
[^6]: KHATIB F, DIMAIO F, COOPER S, et al. Crystal structure of a monomeric retroviral protease solved byprotein folding game players[J/OL]. Nature Structural & Molecular Biology, 2011, 18(10): 1175–1177.http://www.nature.com/articles/nsmb.2119. DOI: 10.1038/nsmb.2119.
[^7]: NOBELPRIZE.ORG. Bruce Merrifield - Facts - NobelPrize.org[EB/OL]. https://www.nobelprize.org/prizes/chemistry/1984/merrifield/facts/.
[^8]: REVIEW C C. Combinatorial Chemistry Review[EB/OL]. http://www.combichemistry.com/.
[^9]: STEINER S, WOLF J, GLATZEL S, et al. Organic synthesis in a modular robotic system driven by a chemical programming language[J/OL]. Science, 2019, 363(6423): eaav2211. http://www.sciencemag.org/lookup/doi/10.1126/science.aav2211.
[^10]: POTYRAILO R A, MIRSKY V M. Combinatorial and High-Throughput Development of Sensing Materials: The First 10 Years[J/OL]. Chemical Reviews, 2008, 108(2): 770–813. https://pubs.acs.org/doi/10.1021/cr068127f.
[^11]: XU C, WU T, DRAIN C M, et al. Effect of Block Length on Solvent Response of Block Copolymer Brushes: Combinatorial Study with Block Copolymer Brush Gradients[J/OL]. Macromolecules, 2006, 39(9): 3359–3364. https://pubs.acs.org/doi/10.1021/ma051405c.
[^12]: MEREDITH J C, KARIM A, AMIS E J. High-Throughput Measurement of Polymer Blend Phase Behavior[J/OL]. Macromolecules, 2000, 33(16): 5760–5762. https://pubs.acs.org/doi/10.1021/ma0004662.
[^13]: GREEN M L, CHOI C L, HATTRICK-SIMPERS J R, et al. Fulfilling the promise of the materials genome initiative with high-throughput experimental methodologies[J/OL]. Applied Physics Reviews, 2017, 4(1):011105. http://aip.scitation.org/doi/10.1063/1.4977487.
[^14]: HATTRICK-SIMPERS J R, ZAKUTAYEV A, BARRON S C, et al. An Inter-Laboratory Study of Zn–Sn–Ti–O Thin Films using High-Throughput Experimental Methods[J/OL]. ACS Combinatorial Science, 2019, 21(5): 350–361. https://pubs.acs.org/doi/10.1021/acscombsci.8b00158.
[^15]: PARDAKHTI M, MOHARRERI E, WANIK D, et al. Machine Learning Using Combined Structural and Chemical Descriptors for Prediction of Methane Adsorption Performance of Metal Organic Frameworks(MOFs)[J/OL]. ACS Combinatorial Science, 2017, 19(10): 640–645. https://pubs.acs.org/doi/10.1021/acs15combsci.7b00056.
[^16]: BROWN F K. Chemoinformatics: What is it and How does it Impact Drug Discovery.[M/OL]//1998: 375–384. https://linkinghub.elsevier.com/retrieve/pii/S0065774308611008. DOI: 10.1016/S0065-7743(08)61100-8.
[^17]: ENGEL T. Basic Overview of Chemoinformatics[J/OL]. Journal of Chemical Information and Modeling, 2006, 46(6): 2267–2277. https://pubs.acs.org/doi/10.1021/ci600234z.
[^18]: 邵学广, 蔡文生. 化学信息学[M]. 北京: 科学出版社, 2013.
[^19]: HAMMETT L P. The Effect of Structure upon the Reactions of Organic Compounds. Benzene Derivatives[J/OL]. Journal of the American Chemical Society, 1937, 59(1): 96–103. https://pubs.acs.org/doi/abs/10.1021/ja01280a022.
[^20]: HANSCH C, FUJITA T. p -σ-π Analysis. A Method for the Correlation of Biological Activity and Chemical Structure[J/OL]. Journal of the American Chemical Society, 1964, 86(8): 1616–1626. https://pubs.acs.org/doi/abs/10.1021/ja01062a035.
[^21]: COREY E J, JORGENSEN W L. Computer-assisted synthetic analysis. Synthetic strategies based on appendages and the use of reconnective transforms[J/OL]. Journal of the American Chemical Society, 1976, 98(1): 189–203. https://pubs.acs.org/doi/abs/10.1021/ja00417a030.
[^22]: COREY E, LONG A, RUBENSTEIN S. Computer-assisted analysis in organic synthesis[J/OL]. Science, 1985, 228(4698): 408–418. http://www.sciencemag.org/cgi/doi/10.1126/science.3838594.
[^23]: 邵学广, 蔡文生. 化学信息学[M]. 第三版. 北京: 科学出版社, 2013.
[^24]: COMOTION U O W. Foldit Standalone | Express Licensing | UW CoMotion[EB/OL]. 2019. https://els.comotion.uw.edu/express_license_technologies/foldit.
[^25]: SciFinder - Explore[DB/OL]. [2019/12/1]. https://scifinder.cas.org/.
[^26]: Royal Society of Chemistry. ChemSpider Search and share chemistry[DB/OL]//Royal Society of Chemistry. 2019[2019-10-23]. http://www.chemspider.com/.
[^27]: CITRINE INFORMATICS. Citrine Informatics[DB/OL]//CITRINE INFORMATICS. https://citrine.io/.
[^28]: Springer Nature. Nature Nano[DB/OL]//Springer Nature. 2019[2019-10-21]. https://nano.nature.com.
[^29]: ELLIOTT J A. Novel approaches to multiscale modelling in materials science[J/OL]. International Materials Reviews, 2011, 56(4): 207–225. http://www.tandfonline.com/doi/full/10.1179/1743280410Y.0000000002.
[^30]: 张伟彬, 杜勇, 彭英彪, 等. 研发硬质合金的集成计算材料工程[J]. 材料科学与工艺, 2016, 24(2): 1–28. DOI: 10.11951/j.issn.1005-0299.20160201.
[^31]: ABRAHAM M, APOSTOLOV R, BARNOUD J, et al. Sharing Data from Molecular Simulations[J/OL]. Journal of Chemical Information and Modeling, 2019, 59(10): 4093–4099. https://pubs.acs.org/doi/10.1021/acs.jcim.9b00665.
[^32]: XU X, RANGE J, GYGLI G, et al. Analysis of Thermophysical Properties of Deep Eutectic Solvents by Data Integration[J/OL]. Journal of Chemical & Engineering Data, 2019: acs.jced.9b00555. https://pubs.acs.org/doi/10.1021/acs.jced.9b00555.
[^33]: 蔡自兴, 刘丽珏, 蔡竞峰, 等. 人工智能及其应用[M]. 第 5 版. 北京: 清华大学出版社, 2016.
[^34]: RACCUGLIA P, ELBERT K C, ADLER P D F, et al. Machine-learning-assisted materials discovery using failed experiments[J/OL]. Nature, 2016, 533(7601): 73–76. http://www.nature.com/articles/nature17439.16 DOI: 10.1038/nature17439.
[^35]: MEYER J G, LIU S, MILLER I J, et al. Learning Drug Functions from Chemical Structures with Convolutional Neural Networks and Random Forests[J/OL]. Journal of Chemical Information and Modeling, 2019, 59(10): 4438–4449. https://pubs.acs.org/doi/10.1021/acs.jcim.9b00236.
[^36]: JOSE R, RAMAKRISHNA S. Materials 4.0: Materials big data enabled materials discovery[M]//Applied Materials Today. 2018. DOI: 10.1016/j.apmt.2017.12.015.
[^37]: C. Mater A, L. Coote M. Deep Learning in Chemistry[J]. Journal of Chemical Information and Modeling, 2019, 59(6): 2545–2559. DOI: 10.1021/acs.jcim.9b00266.
[^38]: GOH G B, HODAS N O, VISHNU A. Deep learning for computational chemistry[J/OL]. Journal of Computational Chemistry, 2017, 38(16): 1291–1307. https://onlinelibrary.wiley.com/doi/full/10.1002/jcc.24764https://onlinelibrary.wiley.com/doi/pdf/10.1002/jcc.24764.
[^39]: materialsproject[EB/OL]. https://www.materialsproject.org/.
[^40]: JAIN A, ONG S P, HAUTIER G, et al. Commentary: The Materials Project: A materials genome approach to accelerating materials innovation[J/OL]. APL Materials, 2013, 1(1): 011002. https://doi.org/10.1063/1.4812323.
[^41]: ONG S P, RICHARDS W D, JAIN A, et al. Python Materials Genomics (pymatgen): A robust, opensource python library for materials analysis[J/OL]. Computational Materials Science, 2013, 68: 314–319. https://linkinghub.elsevier.com/retrieve/pii/S0927025612006295. DOI: 10.1016/j.commatsci.2012.10.028.
[^42]: 国家自然科学基金委员会. 国家自然科学基金委员会-2019 年度项目指南[EB/OL]. 2019. http://www.nsfc.gov.cn/nsfc/cen/xmzn/2019xmzn/js.html.
[^43]: SØRENSEN J J W H, PEDERSEN M K, MUNCH M, et al. Exploring the quantum speed limit with computer games[J/OL]. Nature, 2016, 532(7598): 210–213. http://www.nature.com/articles/nature17620. DOI: 10.1038/nature17620